■ QLS-Scope光片显微镜脊柱3D成像探秘椎间盘微损伤
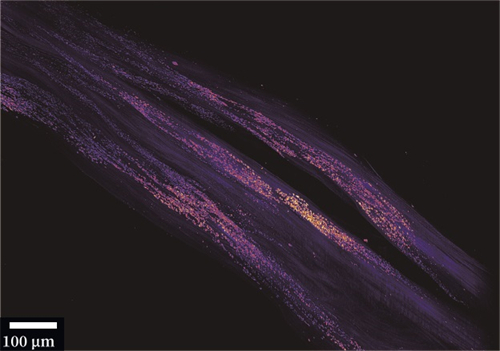
脊柱关键结构如椎间盘和小关节等的损伤常常引起腰疼。到目前为止,准确、无创地检测这些组织中的微损伤仍然非常困难。本文报道了一种基于胶原杂交肽(CHP)的体内成像方法,该方法在分子尺度上专门针对细胞外基质结构的破坏进行标记。利用荧光标记的CHP、活体动物成像和光片荧光显微镜成像,绘制了腰椎胶原蛋白破坏的3D图。
采用PEGASOS方法对小鼠腰椎进行透明化处理,处理后样本在QLS-Scope (Planelight, S.L)上成像。脊柱标本浸泡在BB-PEG培养基中,5倍放大成像,单侧扫描。光片(滤镜组:ex 640 nm, em 661/20 nm)扫描高度设置为0.6 cm, z轴方向步长为3.3 μm。所有图像均采用QScan模式获取,并由QLS软件(Planelight, S.L)进行拼接,保存为16位灰度TIFF图像。使用Imaris软件重建3D图像并制作帧率为25 fps的视频。光片显微成像直观的显示了较相邻正常IVD更强的CHP信号。这表明在未来的临床研究中,CHP具有令人兴奋的潜力。基于CHP的临床成像造影剂和治疗结合剂可用于IVD退行性变和各种脊柱疾病的早期诊断、监测和靶向治疗。
■ QLS-Scope多角度光片成像在小鼠心脏成像中的应用
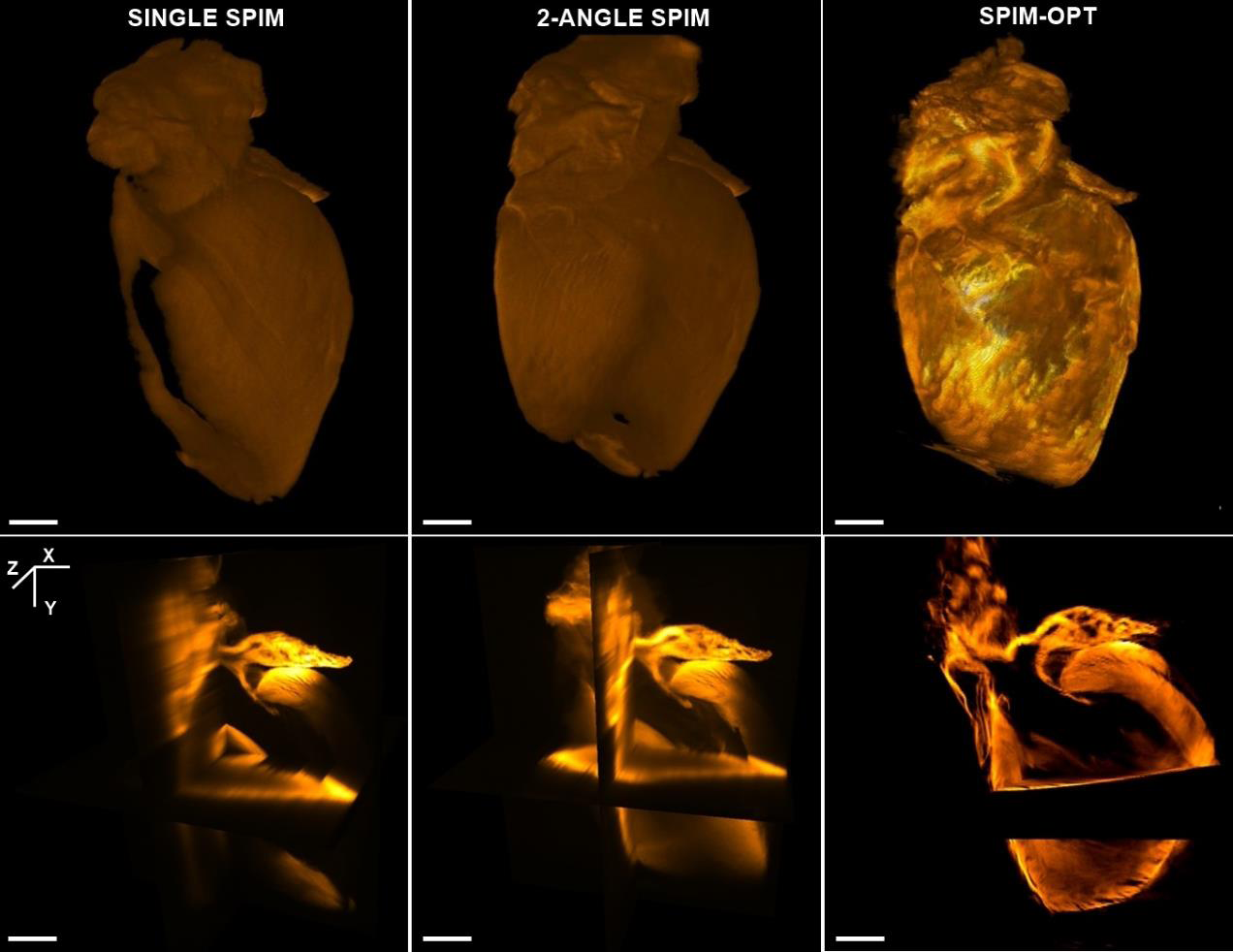
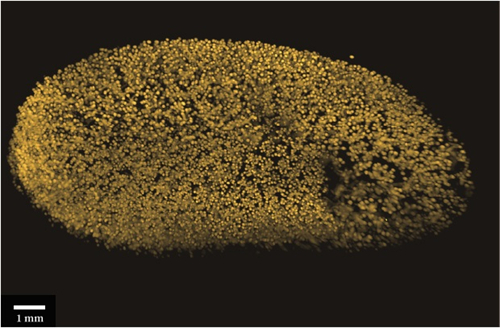
QLS-Scope显微镜具备SPIM-OPT成像模式,通过逐渐旋转样品并在每个层面进行快速SPIM扫描来收集360度光学数据。软件将不同的角度视图重建成一个单一的数据集。这一特性对于从数据中获得具有代表性的3D重建图像非常重要。
如图所示,比较了Single-SPIM、Multiple-SPIM和SPIM-OPT三种方式的成像效果。行显示基于Single-SPIM、Multiple-SPIM和SPIM-OPT三者获得的数据的表面渲染图的背侧面。Single-SPIM成像方式的图例可见,由于缺少来自底层区域的成像信息,终得到渲染不完整图像。Multiple-SPIM成像方式,从另外一个角度集成光学数据在一定程度上可提高图像质量。需要特别注意的是SPIM-OPT成像结果中颜色深度的增加范围大,这样就意味着可以得到更高分辨率的重构图像。SPIM-OPT渲染无空白、无死角,从所有角度成像都可以提取光学数据,即使是透明化程度不是优化的样本也能得到较好的图像效果。第二行显示从相同数据集计算的正交视图。再次证明了,从多个角度进行组合和重建可以显著提高Z分辨率和质量。
QLS-Scope显微镜软件操作的简单性和高扫描速度有助于为每个样本选择和应用合适的成像模式,当您的样本透明化较好时候,可以节约时间的Single-SPIM、Multiple-SPIM等扫描模式;另外,您也可以选择SPIM-OPT模式,以获得更加高Z轴分辨率的高质量的样本图片结果。
图1 Single-SPIM、Multiple-SPIM和SPIM-OPT三种方式的成像效果图(分别为左列、中列、右列)。行显示三种方式成像后表面渲染图的背侧面。第二行显示三种不同模式的样本的正交视图。结果显示,随着软件将重建的附加角度增加,模型的分辨率和清晰度显著的提高了(Scale bars 1 mm)。
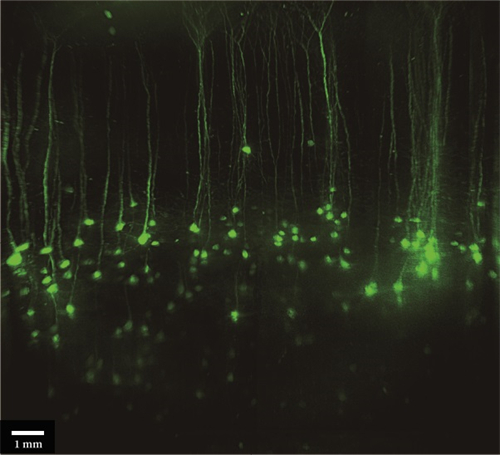
■ QLS-Scope三维成像在拟南芥根系基因表达分析中的应用
光片显微镜的使用可以在不干扰根系生长、破坏无菌或中断实验的情况下实时观察根系。QLS-Scope三维成像具有高分辨率、优化的光学特性。QLS-Scope的高速和简单性来沿着其3D路径跟踪根系生长。下图显示了在生长和沿着中柱表达atHB8驱动的GFP的根。这些测量是在一个体积为2.2*3.1*0.6毫米(4.1毫米3)的单SPIM中进行的,只需45秒即可完成,从而缩短了设备分析和潜在实验改变的时间。此外,在生长培养基中添加稀释的染色剂不会导致背景增加或明显的生长变化。
更多应用案例,请您致电010-85120277/78/79/80 info@qd-china.com